G - PowerPoint PPT Presentation
1 / 41
Title: G
1
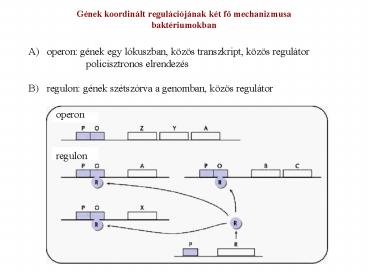
Gének koordinált regulációjának két fo
mechanizmusabaktériumokban
- operon gének egy lókuszban, közös transzkript,
közös regulátor policisztronos elrendezés - regulon gének szétszórva a genomban, közös
regulátor
2
A TRANSZKRIPCIÓS SZABÁLYOZÁS FOBB GLOBÁLIS
STRATÉGIÁI PROKARIÓTÁKBAN
3
A transzkripciós faktorok és aDNS közötti
specifikus kölcsönhatásún. Hélix-Turn-Hélix
(HTH) motívumon megy keresztül
HTH Motívumok
csgD NNEIARSLFISENTVKTH LY
merR IGEVALLCDINPVTLRAWQR
luxR SWDISKILGCSERTVTFHLT
lehet a faktor N vagy C terminálisán, a másik
végen szokott lenni a ligand, kofaktor köto régió
4
BAKTERIÁLIS TRANSZKRIPCIÓS FAKTOROK FOBB
CSALÁDJAI
Faktor család Tagok
AraC család AraC, MelR, RhaS, RhaR, SoxS LysR
család LysR, OxyR, MetR, CysB Crp
család Crp, Fnr MerR család SoxR Két
komponensu NarL, OmpR, Arc szabályozó család
Lac represszor család LacI, GalR MetJ
család MetJ
5
Aktiváció a gén expresszióban I.
Kölcsönhatás - ?CTD-nel (CRP) - ?70 4-es
régiójával (? cI aktivátor) - ?NTD-nel (CRP) - ?
alegységgel (DnaA) - ? alegységgel (N4
single-stranded DNA köto fehérje) - ?CTD-nel és
?70 4-es régiójával (FNR)
Positive activation of gene expression Virgil A
Rhodius, Stephen JW Busby Current Opinion in
Microbiology 1998, 1152-159.
6
Aktiváció a gén expresszióban II.
Promóter konformáció
megváltoztatása - -35 és -10 régió azonos
oldalra kerül (MerR, SoxR) - DNS visszahajlik és
az aktiváló cis szekvencia RNAP fölé kerül - DNS
konformáció változást indukál (FIS, IHF)
Positive activation of gene expression Virgil A
Rhodius, Stephen JW Busby Current Opinion in
Microbiology 1998, 1152-159.
7
Távoli aktivátor helyek segítséget igényelnek
DNS-hajlító fehérje (pl. IHF)
Specifius kötohely
8
Aktiváció a gén expresszióban III.
2 aktivátortól függo promóterek - az aktivátor
kötodése egy másik aktivátortól függ
(eukarióta) - aktivátor kötodése egy másik
aktivátor áthelyezodését eredményezi
(CRP-MalT a malK promóteren) - független
aktiváció (?70 vagy ?NTD és ?CTD) - represszor
müködését gátolja (FIS-NARL/P-FNR)
9
Transzkripció repressziója baktériumokban
10
(No Transcript)
11
Két komponensu szabályozó rendszerek
Komponensek - egy szenzor kináz és egy DNS köto
regulátor - E. coli genom 2 - kb 50 különbözo 2
komponensu rendszer - 3 alcsalád OmpR, FixJ és
NtrC
12
A bakteriális kétkomponensu szabályozó rendszerek
muködése elve
13
OmpR
- OmpR (E. coli)
porin
szervezodés szabályozása ozmózis változás
hatására - általában ?70 használó transzkripciót
aktivál - kölcsönhatás az RNS polimeráz ?
alegységének C terminálisával - ha az N
terminális foszforilálódik megszünik a gátló
hatása a C terminális DNS köto domén felé
14
FixJ
- általában ?70 használó transzkripciót aktivál -
receiver domén deléciója esetén aktív
transzkripció lesz
NtrC
- N terminális receiver és C terminális DNS köto
domén között egy központi ATPáz domén (glicin
gazdag Walker box) - DNS köto domén FIS-hez
homológ (FIS eukarióta enhancer köto
fehérje) - általában ?54 használó transzkripciót
aktivál
15
ArcA/B
- aerobic respiratory control - ArcB (szenzor
kináz) sejt redox és metabolikus helyzet
(elektron transzport változást érzékel) -
ArcA(citoplazmatikus regulátor) ArcB
foszforilálja ? aktív - pO2 1-5 mbar között -
TATTTaa konszenzus szekvencia - Haemophilus ArcA
- E. coli homológ gén OmpR
ArcA
ArcB
O2
P
ArcA?P
16
Az ArcAB és FNR anaerob aktivációja
17
NarX/L és NarP/Q
- nitrát regulator - NarX és NarQ membrán
szenzor kináz - NarL és NarP citoplazmatikus
regulátor - szignál nitrát és nitrit - nitrát
metabolizmusra hat - NarL és NarP különbözo
génekre különbözo atás ? a génexpresszió
finom hangolása
Kétkomponensu rendszerekvége
18
A lac operon kettos szabályozása
- laktóz (allolaktóz) indukál
- glükóz gátol, cAMP/CAP-n
- keresztül
- glükóz/egyéb cukor kiiktatása tápból nem
célszeru - ? glükóz szabályozás kikapcsolása
19
lac (és trp) alapú promóterek
erosség
-35 -10
trp AATGAGCTGTTGACAATTAATCATCGAACTAGTTAACTAGTACG
CA tac AATGAGCTGTTGACAATTAATCATCCGGCTCGTATAATGT
GTGGA lacUV5 CCCCAGGCTTTACACTTTATGCTTCCGGCTCGTAT
AATGTGTGGA lac CCCCAGGCTTTACACTTTATGCTTCCGGCTCGT
ATGGTGTGTGGA
20
A transzkripciós faktorok sokoldalúak.
21
Transzkripció termináció baktériumokban
Rho független
Rho függo
22
A transzkripció és a transzláció
párhuzamosanmegy baktériumokban
23
(No Transcript)
24
A triptofán operon szabályozása
25
Termináció attenuáció trp operon
26
Termináció - antitermináció
túl sokat nem lehet vele kezdeni, génen belüli
sajátság
27
E. coli-ban fehérje túltermeltetésre használt
promóterek
28
mRNS degradáció baktériumokban
mRNS stabilitás prokariótákban néhány perc,
eukariótákban órás nagyságrend elobb utóbb
minden RNS lebomlik mRNS stabilitását
meghatározó faktorok - belso, saját
szerkezet - a környezet hatására bekövetkezett
változás a degradációs apparátusban puf operon
(a fotoszintetikus komplex komponensei)
Rhodobacter capsulatus degradációja O2 hatására
felgyorsul policisztronos rendszerek esetén az
alegységek arányának szabályozása a mRNS
régióinak eltéro stabilitásával
29
A R. capsulatus puf mRNS régióinak stabilitása
30
mRNS degradáció baktériumokban, vizsgálati
módszerek
- - transzkripció gátlása (pl. rifampicin) t0
idopontban, - majd ?? idoközönként mintavétel és RNS analízis
(Northern..) - a degradációs mechanizmusban szerepet játszó
gének deléciója, - homérséklet érzékeny expressziós változatának
kialakítása - - in vitro transzkripció jelölt nukleotidokkal,
a kapott termék inkubációja a sejtextraktummal
különbözo ideig, majd analízis
gélelektroforézissel, kvantitálás
31
RNázok, RNS degradáció
Bacterial exoribonucleases Polynucleotide
phosphorylase Ribonuclease PH Ribonuclease
II Ribonuclease R RNase D Ribonuclease
T Ribonuclease BN Oligoribonuclease
Bacterial endoribonucleases Ribonuclease
I Ribonuclease III Ribonuclease P Ribonuclease
E Ribonuclease HI
A degradáció iránya virtuálisan 5' ? 3' irányú,
de ilyen enzimaktivitást nem lehet kimutatni
prokariótákban Megoldás kombinált enzimmuködés ?
degradoszóma
32
mRNS-t stabilizáló-destabilizáló tényezok
33
Az 5 végi struktúra stabilizáló hatása
- a stabillizálódás a mRNS hurok struktúrájában van
- nem a riboszóma véd,
- a stabilizáló effektus átviheto más génekre
34
mRNS-eket stabilizáló (védo) tényezok
- 5 végi trifoszfát
- RNS struktúra
- riboszóma
35
A degradoszóma felépítése
36
Az RNaseE elsodleges felépítése
Érdekes módon sok bakteriális genomban nincs meg
37
A degradoszóma komponensei I.
Endoribonuclease E (RNáz E)
- 1061 aminosav ? 118 kDa fehérje, virtuálisan 180
kDa (oka prolin gazdag régió) - felismero hely
- (A/G)AUU(A/T)
- vagy egy komplex másodlagos struktúra
- 5' monofoszfátot preferál? 5' trifoszfát
stabilizál
N-terminális régió (50 kDa) hasonlít a Caf-re
(cytoplasmic axial filament protein) ?
feltételezett funkció a belso RNS transzportban N
terminális 70 aa (S1 domén) hasonlít a PNPase és
RnaseII (illetve (CSP, cold shock protein, RNS
chaperon) RNS köto doménjére C-terminálisa
degradoszóma egyéb komponenseire megfelelo köto
domének
38
A degradoszóma komponensei II.
- PNPase (polynucleotide phosphorylase)
- 78 kDa alegységek, homotrimer
- 3' ? 5' foszfát függo processzív exonukleáz,
- ribonukleotid difoszfátok képzodnek
- poliadenilációs aktivitás
- Polyphosphate Kinase (PPK)
- funkció ATP regeneráció,
- polifoszfát (inhibiálja a degradációt)
eltávolítás - ppk mínusz törzs megnövekedett mRNS stabilitás
- 80 kDa alegységek, homotetramer, sok van E.
coli-ban
39
A degradoszóma komponensei III.
- Helikáz
- ATP függo DEAD (Asp-Glu-Ala-Asp) helikáz
- 50 kDa RhlB
- a másodlagos struktúrák kinyitása szétroncsolása
40
Egyéb mRNS degradációjában résztvevo enzimek
- RNáz II
- 70 kDa monomer,
- a sejt 3' ? 5' exoribonukláz aktivitásának 90-a
- ribonukleotid monofoszfátok képzodnek
- a PNPáz-zal együttes deléciója letális !!!
- PolyA polimerázok
- PAPI 53 kDa
- PAPII 35 kDa
- poliadeniláció, 15- 50 bázis hosszú
- mRNS instabilitás
41
A mRNS degradáció mechanizmusa